PI介绍-吴健民

研究员、博士生导师
北京大学生物信息中心博士研究生毕业(2006),芬兰赫尔辛基大学博士后(2006-2010),澳大利亚悉尼Garvan医学研究所研究组长(2010-2015)和新南威尔士大学兼职高级讲师(2012-2015)。现北京大学肿瘤医院/研究所肿瘤生物信息中心主任。在癌症基因组和蛋白组相关的生物信息学领域开展了系列研究,获得澳大利亚NHMRC、Cancer Council NSW和Vodafone Foundation等多个机构的科研资助。以第一作者/通讯作者在《Nature Reviews Cancer》、《Nature Methods》、《Nucleic Acids Research》等杂志发表论文。
电话:010-88196986(办公室)
邮箱:wujm@bjmu.edu.cn
主要研究领域
主要研究方向:1)综合生物信息计算和实验手段,整合基因组和蛋白组研究来深入了解胃癌等国内高发癌种的异质性和分子分型;2)在分子网络层次理解肿瘤分子变异的信号通路和功能;3)多瘤种的分子和医学大数据的深度挖掘和整合分析。
代表性研究成果
1. 生物信息分析工具和平台的开发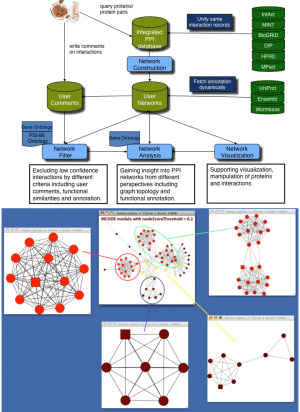
开发了一系列生物信息分析平台,可应用于组学结果的分析,其中包括:1)用于KEGG信号通路富集分析的KOBAS在线服务器(Nucl Acids Res 2006);2)生物信息分析流程平台WebLab (Nucl Acids Res 2009);3)集蛋白作用网络构建、过滤、分析和可视化工具一体的分析平台PINA(Nat Methods 2009);4)PINA第二版本(Nucleic Acids Res 2012)加入了蛋白作用网络模块的识别、功能注释和富集分析。
2. 癌症组学研究
负责国际胰腺癌基因组项目(ICGC Pancreatic Cancer)中多组学数据的功能分析、临床数据关联和整合分析,在ICGC胰腺癌队列的外显子组(Nature 2012)、全基因组(Nature 2015)和转录组(Nature 2016)研究以及分子机制研究(Cell 2014;Gut 2014)中都有贡献,为发现相关癌症驱动基因和信号通路的异常提供了重要线索,并发现了具有临床应用转化价值的若干分子标志物和新的分子分型。领导的食道腺癌的基因组研究(J Med Genet 2016),发现人乳头瘤病毒阳性和阴性的病人具有不同的突变特征:病毒阳性者体细胞突变/癌症驱动基因突变更少。
与Monash大学蛋白磷酸化专家Roger Daly教授合作开展了一系列的肿瘤蛋白组和磷酸化组研究,发现磷酸化特征和相关的蛋白信号网络不仅可以用来进行肿瘤分子分型(Breast Cancer Res 2014),还可以识别PDX细胞株亚群的靶向药物敏感性(Mol Cell Proteomics 2016);通过对公开基因组学数据的整合,对癌症驱动基因中的蛋白激酶进行了首次多癌种的分析和归类,并结合蛋白组数据和功能基因组数据阐述了特定激酶对遗传背景的依赖性,以及各种组学研究的互补性(Nat Rev Cancer 2016)。
代表性论文
1. Fleuren ED, Zhang L, Wu J*, Daly RJ* (2016) The kinome “at large” in cancer. Nat Rev Cancer 16: 83-98
2. Humphrey ES, Su SP, Nagrial AM, Hochgräfe F, Pajic M, Lehrbach GM, Parton RG, Yap AS, Horvath LG, Chang DK, Biankin AV, Wu J*, Daly RJ* (2016) Resolution of novel pancreatic ductal adenocarcinoma subtypes by global phosphotyrosine profiling. Mol Cell Proteomics (in press)
3. Rajendra S*, Wang B*, Merrett N, Sharma P, Humphris J, Lee HC, Wu J* (2016) Genomic analysis of HPV-positive versus HPV-negative oesophageal adenocarcinoma identifies a differential mutational landscape. J Med Genet 53(4): 227-31
4. Ali NA#, Wu J#, Hochgräfe F#, Chan H, Nair R, Ye S, Zhang L, Lyons RJ, Pinese M, Lee HC, Armstrong N, Ormandy CJ, Clark SJ, Swarbrick A, Daly RJ (2014) Profiling the tyrosine phosphoproteome of different mouse mammary tumour models reveals distinct, model-specific signalling networks and conserved oncogenic pathways. Breast Cancer Res 16(5): 437
5. Cowley MJ, Pinese M, Kassahn KS, Waddell N, Pearson JV, Grimmond SM, Biankin AV, Hautaniemi S and Wu J* (2012) PINA v2.0: mining interactome modules. Nucl Acids Res 40: D862-5
6. Wu J*, Vallenius T, Ovaska K, Westermarck J, Makela TP, Hautaniemi S. (2009) Integrated network analysis platform for protein-protein interactions. Nat Methods 6: 75-7
7. Liu X#, Wu J#, Wang J, Liu X, Zhao S, Li Z, Kong L, Gu X, Luo J and Gao G. (2009) WebLab: a data-centric, knowledge-sharing bioinformatic platform. Nucl Acids Res 37, W33-39
8. Wu J#, Mao X#, Cai T, Luo J and Wei L. (2006) KOBAS server: a web-based platform for automated annotation and pathway identification. Nucl Acids Res 34, W720-724
实验室成员

研究人员:崔思佳、杜阳、张潞西 技师人员:邢丽影














